Logiciels et applications digitales - Agriculture durable
- Accueil
- Collaborer avec nous
- Solutions Cirad'Innov
- Produits et services
- Gigwa : Système de gestion des données de génotypage
Gigwa : Système de gestion des données de génotypage haut débit
Giwa © Cirad
L’analyse simplifiée de grands volumes de données génomiques
Comment mieux évaluer et utiliser la biodiversité des plantes cultivées dans les pays tropicaux ? Telle était l’ambition du projet européen ARCAD dont est issue la solution Gigwa. Né de l’expertise du Cirad et de l’IRD en bio-informatique, le système visa en premier lieu à faciliter la centralisation et la manipulation des grands volumes de données génotypiques issus de l’émergence du séquençage NGS. Ses dernières adaptations permettent désormais le partage, la visualisation et l’analyse de ces données. Cet outil est en évolution constante afin de servir le plus grand nombre d’applications possibles pour les différents secteurs de l’agronomie et de la santé.
Doté d’une interface web ergonomique, capable de s’adapter aussi bien aux données animales qu’humaines ou végétales, et prenant en charge aussi bien les métadonnées associées au matériel biologique que les annotations fonctionnelles des variants, Gigwa est une solution et qui se prête à de nombreux cas d’utilisations. Il peut en effet aussi bien faire office d’entrepôt de données que de convertisseur entre divers formats, d’outil de filtrage, visualisation et analyse en ligne, ou de source de données interopérables dans des workflows de tous types.
Avec l’appui de notre équipe de développement, le système peut être intégré à d’autres plateformes web (e.g., Genome-Hubs, PeanutBase, Musa Germplasm Information System, Breeding Management System), et des fonctionnalités complémentaires peuvent être développées à la demande des utilisateurs.
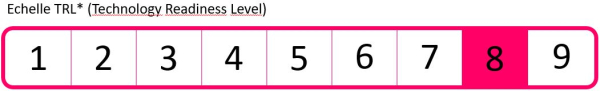
Stade de développement
Les atouts de la solution Gigwa
Gigwa, Genotype Investigator for Genome-Wide Analysis, offre un large éventail de services. Ainsi, la plateforme :
- gère des dizaines de milliards de génotypes ;
- s’installe facilement sur un poste local ou sur un serveur, via une image Docker ;
- peut être déployé en mode multi-utilisateurs, gérant l’authentification unique (SSO) pour un usage institutionnel facilité ;
- est interopérable à travers divers standards d’APIs REST (BrAPI v1 & V2, GA4GH)
- permet de nombreux formats d’import et d’export de données ;
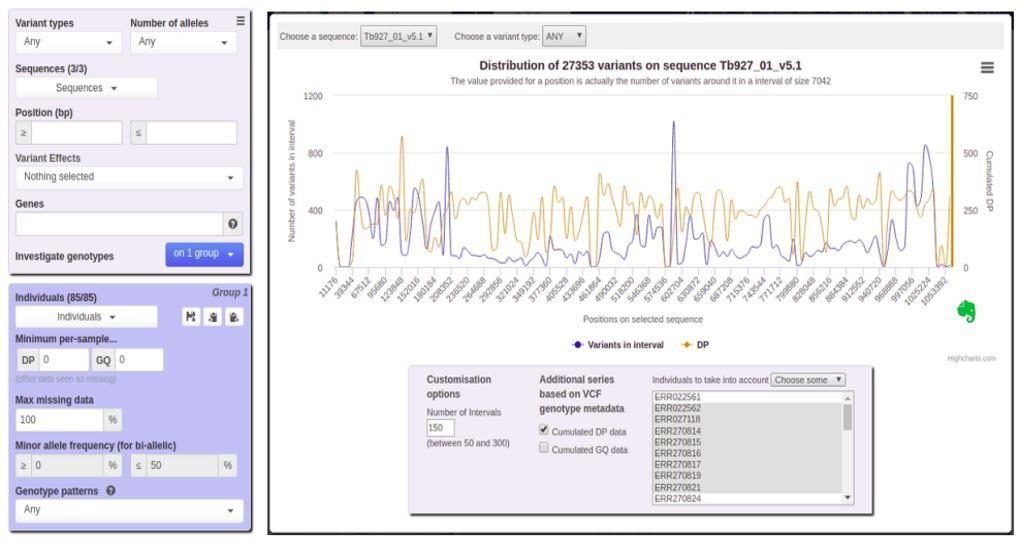
- offre le filtrage de caractéristiques des variants (position, annotations…), sur la base du polymorphisme entre individus ou sur des critères de qualité des données ;
- accepte différents niveaux de ploïdie et prend en compte les données phasées
- archive des critères de recherche sous forme de favoris ;
- présente une fonctionnalité de recherche de variants discriminants entre 2 groupes d’individus
- permet la représentation graphique de statistiques calculées en temps réel (distribution des variants, MAF, Fst, Tajima’s D) ;
- intègre des outils de visualisation et d’analyse intégrés (IGV.js, Flapjack-Bytes) ;
- facilite la connexion avec des outils extérieurs pour des analyses plus poussées (e.g., Galaxy, SniPlay...) ;
- inclut une fonctionnalité de sauvegarde et de restauration des bases de données pour une meilleure sécurisation des données critiques
Un outil en appui des acteurs de l’analyse de données de séquençage
Au service de la génomique appliquée à la sélection et au diagnostic variétal ou animal
Les fonctionnalités de Gigwa peuvent répondre à une large gamme des besoins des entreprises évoluant dans le domaine de l’analyse des données génomiques. L’outil étant assez intuitif, il permet aux chercheurs de prendre l’outil en main sans forcément recourir à un bioinformaticien. Comparativement aux autres outils open source et gratuits du marché, il propose des applications complémentaires, notamment la possibilité de comparer 2 groupes d’individus sur la base de critères d’intérêt.
L’ouverture du système (code en open source et interopérabilité au travers d’APIs) constitue également un intérêt pour les sociétés disposant d’une solution internalisée, pour laquelle Gigwa peut apporter des fonctionnalités complémentaires et servir de plateforme pour le développement de modules répondant à des besoins spécifiques.
Au service de l’enseignement en matière de génétique et génomique
Nombre d’outils open-source existent, mais n’offrent ni la puissance de Gigwa, ni sa simplicité d’utilisation. Avec plus de 40 ans d’expertise dans l’étude génomique des plantes tropicales et méditerranéennes, nous avons à cœur d’accompagner les structures de formation et d’enseignement avec des outils utiles et performants. Ainsi, Gigwa est actuellement utilisé comme support de travail pour des études de cas soumises à des étudiants de Master 2 de l’Université de Montpellier, Dakar et Antananarivo dans le cadre d’un module dispensé par des chercheurs et ingénieurs du CIRAD.
Comment obtenir les services de Gigwa ?
Une instance de Gigwa hébergée par le Cirad est en accès libre à l’adresse suivante https://gigwa.southgreen.fr/gigwa/
Si vous souhaitez vous rendre compte du mode de fonctionnement de l’outil, 4 vidéos de démonstration sont à votre disposition :
- Importez vos données sur la plateforme ;
- Filtrez vos données ;
- Exploitez vos données filtrées ;
- Discriminez les variants en fonction de phénotypes d’intérêt.
Gigwa vous intéresse et vous souhaitez étudier la possibilité d’utiliser l’outil pour gérer vos propres données de variations génomiques, l’intégrer à votre système d’information ou développer des modules complémentaires ? Contactez-nous : ciradinnov@cirad.fr
La communauté de développeurs et d’utilisateurs réguliers contribuant à l’évolution du système Gigwa associe à l’échelle locale 3 unités.
- INTERTRYP, engagée dans la lutte contre les maladies tropicales négligées dues aux Trypanosomatidés pour améliorer la santé humaine et animale,
- AGAP Institut qui développe des recherches sur l’amélioration génétique et l’adaptation des plantes tropicales et méditerranéennes,
- DIADE qui œuvre pour la préservation des écosystèmes tropicaux et la valorisation de la biodiversité, mais aussi des partenaires internationaux : Bioversity International, Integrated Breeding Platform, ICARDA.
Références et propriété intellectuelle
Code source
Intégralement disponible sous licence GNU Affero General Public License v3.0 à l’adresse suivante https://github.com/SouthGreenPlatform/Gigwa2
Dépôt à l’Agence Protectrice des Programmes réalisé le 05/04/2019 sous le numéro IDDN1 .FR2 .0013 .1500364 .0005 .S6 .C7 .20198 .0009 .2090010
Publications
Managing high-density genotyping data with Gigwa. Sempere Guilhem, Larmande Pierre, Rouard Mathieu. 2022. In : Plant Bioinformatics: methods and protocols. Edwards David (ed.), 415-427. (Methods in Molecular Biology, 2443) ISBN 978-1-0716-2067-0
https://doi.org/10.1007/978-1-0716-2067-0_21
Gigwa v2—Extended and improved genotype investigator. Sempere Guilhem, Petel Adrien, Rouard Mathieu, Frouin Julien, Hueber Yann, De Bellis Fabien, Larmande Pierre. 2019. GigaScience, 8 (5), 9 p.
https://doi.org/10.1093/gigascience/giz051
Gigwa—Genotype investigator for genome-wide analyses. Sempere Guilhem, Philippe Florian, Dereeper Alexis, Ruiz Manuel, Sarah Gautier, Larmande Pierre. 2016. GigaScience, 5 (25), 9 p.